Quantificació relativa normalitzada respecte un gen de referènciaHabitualment, la quantificació relativa s'aplica per conèixer si l'expressió d'un gen específic varia entre dos grups, per exemple: grup control vs grup tractat.Per obtenir una quantificació relativa que s'ajusti al màxim a la realitat, les dades obtingudes de la Real-time RT-PCR s'han de normalitzar. Seguir unes estartègies de normalització apropiades permetrà controlar possibles errors introduits al llarg del procés experimental. En la quantificació relativa podem diferenciar entre dos mètodes de normalització:
Gen de referènciaEl control intern o gen de referència es caracteritza per presentar una expressió constant i invariable en totes les mostres. L'expressió d'aquest no pot ser modificada per les condicions de l'estudi o per la resposta a un tractament experimental. La literatura ens mostra que no s'han identificat gens de referència òptims (els gens utilitzats habitualment varien considerablement entre mostres), no obstant aquest mètode de normalització és el més utilitzat. Per aquest motiu, l'eina desenvolupada implementa un mètode per tal d'identificar els millors gens control. El mètode utilitzat és el conegut com Vandesompele. El mètode Vandesompele es basa en l'estabilitat dels gens per identificar el gen de referència, com més estable sigui un gen, millor gen de referència serà.Així, s'obté un ranking amb els gens més estables, permeten l'ús de més d'un gen de referència si es dóna el cas (es recomana utilitzar 2 gens de referència per fer la normalització). Un cop identificats els gens amb la millor estabilitat es realitza la mitja geomètrica d'aquests amb la que es normalitzaran les dades. Quantifiació relativaUn cop s'ha seleccionat el gen de referència té lloc la quantificació relativa.Per calcular l'expressió relativa a un gen de referència disposem de tres mètodes diferents: 2-ΔΔCT Method, 2-ΔCT Method i Pfaffl Method. L'eina CT utilitza una fórmula matemàtica derivada del Pfaffl Method pel càlcul de l'expressió relativa (aquesta és similar al mètode presentat per Soong et al. -Roche Diagnostics (2000)-). L'algoritme matemàtic derivat del Pfaffl method utilitzat es defineix com: 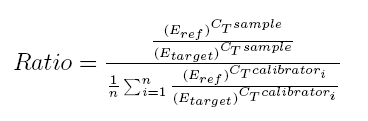 Respecte a altres mètodes aquest permet el càlcul de l'expressió relativa tenint en compte l'eficiència de cada gen i utilitzant més d'una mostra calibradora. Eficiència de la Real-time RT-PCRDe forma general, la PCR consisteix en quatre etapes diferents, similars a la cinètica de creixement bacterià observada per Monod. En la segona etapa de la cinètica de Monod té lloc la fase logarítmica, equivalent al creixement exponencial del producte de la PCR --idealment la D.O. del producte de PCR es dobla en cada cicle-- mesurat com a senyal de fluorescència. La cinètica de la PCR en aquesta fase pot descriures com:D'aquesta manera, el càlcul de l'eficiència per un gen específic s'obté de: Eff=10slope. S'ha descrit en la literatura com l'ús, per defecte, de valors d'efficiència de 2 (100% de l'eficiència de la PCR) afecta significativament al càlcul acurat dels nivells d'expressió relatius a un gen específic; observant fins a un error de 4-fold quan l'eficiència varia tant sols en un rang de 0,04. Per calcular l'eficiència de cada gen, l'eina CT utilitza les dades de fluorescència mesurades per cada cicle al llarg de tot el procés d'amplificació. L'algoritme implementat pel càlcul de l'eficiència es basa en el programa LinRegPCR. Concretament, el procés per obtenir l'eficiència consisteix en linearitzar les dades de la fase exponencial de la PCR i determinar la millor regressió lineal. Aquest procés es dur a terme per cada un dels gens i cada mostra, obtenint un resultat específic per cada cas. Els valors finals d'eficiència per gen són el resultat de la mitja dels diferents valors de cada gen. 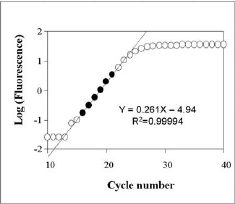
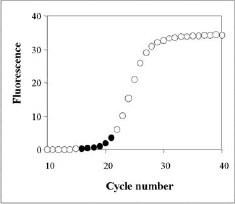
En les dos figures que es mostren a la dreta es representa el funcionament del programa per obtenir el valor de l'eficiència. |
||





